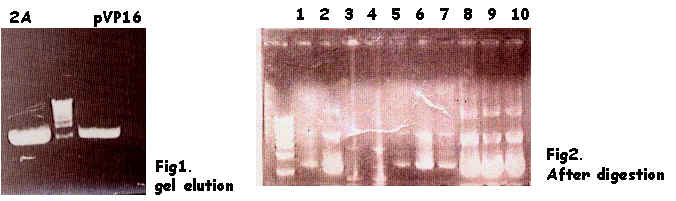
pVP16/2A

將 pM/2A 和 pVP16 double digestion 後,Ligation, Transform ,Transform 除了 pVP16/2A外,另外也做了 pM only with ligase(可知Cip是否完全)以及pM without ligase(可知pM是否有切完全) 。之後Purification of Plasmid DNA,發現2,8,9的濃度較高,經過 digestion 確認後,將這三株送 sequence.得到正確的結果,Inframe也正確。
pM/PKR
PCR
buf×10 5λ Taq 1λ
5'(20mol) 5λ H2O 33λ
3'(5μM) 5λ Templet 1λ
dNTP 1λ
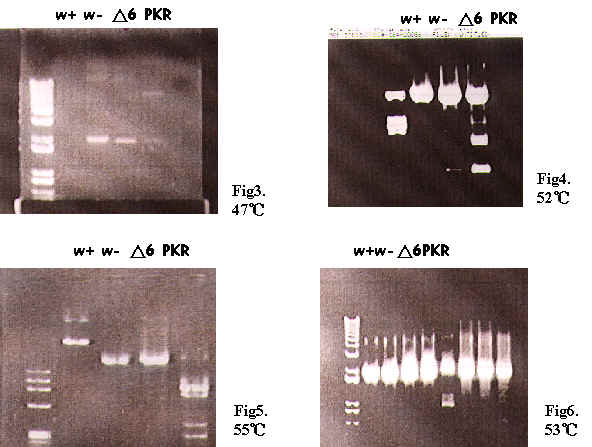
annealing temperature 47℃時,w+,PKR 的濃度不高,所以升高溫度,直到55℃時,primer 變得亂黏。所以再將溫度降回53℃,之後所做的 PCR都是用這個溫度。送出去的 sequence 也確定是正確的。
pM cloning
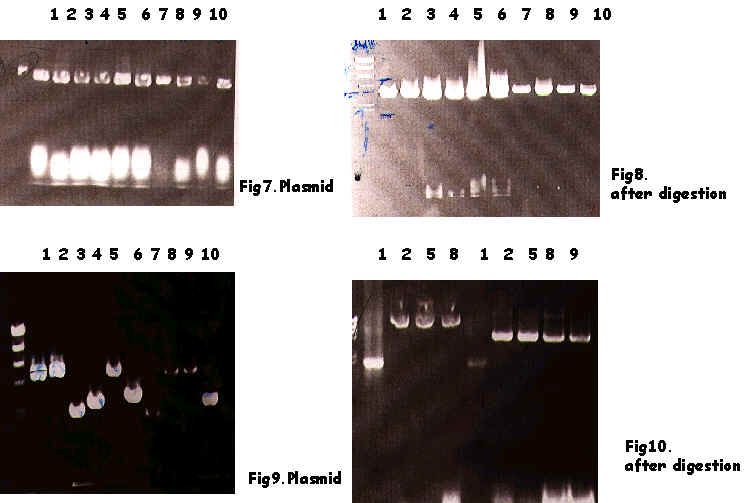
pM 從 plate 上 extraction出來,送 sequence確定後,將 PKR 放入 pM中。實驗反覆做了好幾次,但是PKR始終無法放入pM當中 , 有時plasmid看起來就都是空的,切出來的大小和沒切根本差不多 。有時候plasmid看起來好像有東西,band 高高低低的,切過後雖然 size 有變小,但仍然沒切出 PKR ,應該只是從 circular 切成 linear而已, 所以還是沒有 insert。
EcoR1 digestion
因為懷疑 BamH1 or Xba1 可能失去作用,故用 EcoR1 digestion試試是否有 insert。理論上如果有 insert,兩個 EcoR1 site會相差約350 bp。
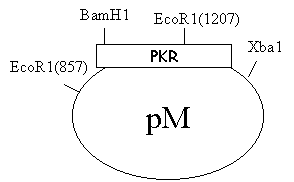
Fig.
11 EcoR1
EcoR1確實有在底下切出一條小小的 band,而且 size也有可能是300bp,所以將這兩支送sequence。但結果仍然不是,甚至根本讀不出 signal。
TA cloning (PCR3.1)
因為 BamH1 and Xba1 site 在 PKR的兩端,所以考慮到可能太接近邊也至於 enzyme 無法作用,所以先將 PKR接到 PCR3.1上,之後再digestion。
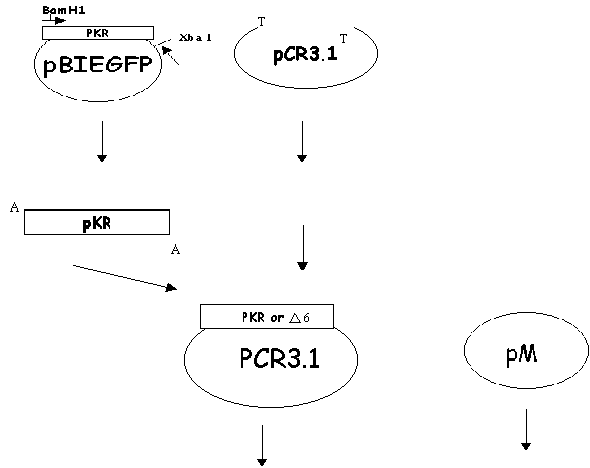
Double digestion ( BamH1 + Xba1 )
![]()
![]()
Cip treatment
![]()
![]()
![]()
![]()
![]()
PKR △6

1 2 3 4 5 6 7 8 9 10 1 2 3 4 5 6
Fig 12. PCR3.1/PKR
plasmid
PKR △6
1 2 3 4 5 6 7 8 9 1 0 1 2 3 4 5 6
Fig
13.after digestion
和 pM的結果類似,雖然切過後 size 變小,但仍然切不出 PKR。
pGEMT cloning
因為 pGEMT上有一段 sequence可使 colonies 產生淡藍色,所以將 pGEMT 放入insert後會改變 sequence,失去藍色的基因 。相反的如果 self ligation則會有藍色表現,因此要挑 plate 上白色的 colony,應該就是有insert的 plasmid 。
Fig 14. GEMT/PKR
Fig 15.after digestion
結果雖然挑白色的 colonies,但仍然沒有 insert。理由可能是因為即使沒有insert,pGEMT的 sequence 有時候仍不會100%表現,因為 sequence偶而會發生錯誤,或是transcription or translation發生錯誤 。
嘗試了許多方式後仍然沒有辦法取得
pM/PKR plasmid,
pM + BamH1 pM + Xba1
![]()
![]()
![]()
![]()
+ Xba1 + BamH1 enzyme work
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]() ligase
Cip and
ligase
Cip and
ligase + - + - + - + - pM digestion work
![]()
transform
另外第九個 sample 是 no DNA,觀察 competent cell 是否已受污染。
透過這些過程,希望從中能尋找出究竟問題出在哪,因為理論上所有條件都具備了,沒有理由 insert 會放不進去。但很可惜的是實驗並不來得及做完,最終還是不知道問題出在哪就要離開實驗室了。